개요[편집 | 원본 편집]
전사 과정에서 나온 pre-mRNA를 성숙한 mRNA로 만드는 과정 중 하나. 이 과정을 통해 인트론이 제거되고 엑손끼리만 결합하게 된다. 원핵생물에서는 거의 일어나지 않고 진핵생물에서 주로 일어난다. RNA editing과는 다르다.
DNA에는 인트론(intron)과 엑손(Exon)이 있다. 인트론은 단백질에 대한 정보가 없는 non-coding 서열이고, 엑손은 coding 서열이다. 원핵생물은 인트론이 거의 없지만[1], 진핵생물은 엑손 사이에 수많은 인트론들이 끼어들어가 있다. 하지만 RNA 중합효소는 엑손과 인트론을 구분하지 않고 모조리 RNA로 전사하는데, 때문에 원하는 단백질을 만들기 위해서는 반드시 RNA 이어맞추기(splicing)을 통해 인트론을 제거해야 한다. 위의 그림자료 처럼 이어맞추기가 완료되어도 5' cap과 poly-A가 남아있는데, 엑손으로 취급 받기 때문이다. 이 둘은 특이하게도 non-coding 엑손으로 불린다.
고등생물로 갈 수록 인트론의 수가 늘기 때문에 인트론과 엑손을 정확히 구분하는 것이 매우 중요해진다. 사람의 경우는 전체 유전자 중 10% 내외만 엑손인 것으로 알려져있다. 진핵생물은 이어맞추기 복합체(spliceosome)이라는 특수한 단백질이 전문적으로 이어맞추기를 한다.
발견[편집 | 원본 편집]
20세기 중후반, 핵에 존재하는 mRNA가 세포질에 있는 mRNA보다 훨씬 길다는 사실은 유전학의 수수께끼 중 하나였다. 그러던 중 1977년 Richard J. Roberts와 Phillip A. Sharp는 각각 아데노바이러스(adenovirus) 연구를 통해 인트론[2][3]의 존재와 RNA splicing을 밝혀냈고, 당시 전사에 대해 갓 이해하고 있던 생명과학자들에게 큰 충격을 가져다주었다. 결국 두 과학자는 1993년 노벨생리학상을 받게된다.[4]
아데노바이러스는 대표적인 감기 바이러스로, 당시 진핵생물의 유전적 이해를 위해 실험에 사용되고 있었다. 아데노바이러스 안에는 하나의 DNA가 있고, 이 DNA 상에 아데노바이러스를 구성하는 단백질들이 코딩되어 있다. 처음에는 아데노바이러스 각 단백질들의 mRNA 5 말단을 조사해서 전사 개시 서열을 추측해 게놈 지도를 만드려는 실험이었다. mRNA에 DNA를 혼성결합(hybridizaiton) 시켜서 DNA의 어느 지점이 결합되는지를 확인하는 것이 실험 과정이었다. 그런데 이상한 일이 일어났다. mRNA에서는 서로 연속적이던 5 말단 서열이, DNA상으로는 따로 떨어져있었던 것이다. Roberts는 서로 분리(split)되어 있는 개시서열을 3군데 찾아냈고, 이를 tripartite leader라고 이름을 붙였다. 즉 DNA 상에 흩어져있는 3개의 leader들이 mRNA에서는 알 수없는 방식으로 합쳐져있다는 것이 밝혀진 것이다. 실험을 더 진행시켜보니, 똑같은 tripartite leader를 가졌는데 뒤에 오는 단백질 서열만 다른 mRNA들이 발견되었고, 이는 결국 인트론과 RNA 이어맞추기의 발견으로 이어지게 되었다.
그림자료 만들기 귀찮으므로 직접 설명하는 영상을 보도록 하자. [2]
매커니즘[편집 | 원본 편집]
인트론 - 엑손 경계[편집 | 원본 편집]
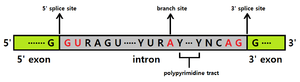
인트론과 엑손은 그 경계에 특정 서열을 가지고 있다.[5][6] 이 서열들은 대부분의 인트론, 엑손에서 공통되는 서열들로, 5' splice site(혹은 donor site), branch site, 3' splice site(혹은 acceptor site) 총 세 부분으로 나뉜다. 5' splice site는 인트론의 5' 말단을 의미하고, 3' splice site는 인트론의 3' 말단을 의미한다. branch site는 인트론의 중간에 위치하는데, 엄밀히 따지면 3' 말단에 더 가까이 있고 뒤로 폴리피리미딘 서열이 뒤따른다. 5' splice site의 GU 서열, 3' splice site의 AG 서열, branch site의 A 서열은 모든 진핵생물에서 가장 잘 보존된 서열이며 인트론에 반드시 포함되어있다. 이와 반대로 엑손은 단백질 정보를 담아야하기 때문에 덜 보존되어있다.
참고로 그림자료에서 R은 퓨린(A,G) 중 하나, Y는 피리미딘(C,T) 중 하나, N은 아무 염기 중 하나를 의미한다.
이어맞추기 반응[편집 | 원본 편집]
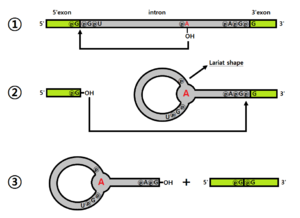
인트론은 두번의 transesterification(에스테르 전이반응)에 의해 제거된다. branch site 아데노신(A)의 2'-OH(nucleophile)가 spliceosome(후술)의 도움을 받아 5' splice site 구아닌(G)의 인산기를 공격한다. 이렇게 G와 A가 인산디에스테르 결합으로 연결되어, 밧줄 매듭 모양의 루프를 만들게 된다. 이것을 'Lariat shape'라고 하고, A를 중심으로 세갈래길(3 way junction)이 만들어진다[7]. 그리고 G-A 연결에 의해 인트론 5'말단 쪽에 있던 exon이 절단된다.
절단된 5' 엑손의 3'-OH기는 인트론의 비스한 방식으로 3' splice site를 절단하고 결합한다. 이렇게 인트론 루프와 조합된 엑손이 생기고, 이어맞추기는 끝나게 된다.
만약 mRNA가 두개 있다면, 이들 사이에서도 이어맞추기가 일어날 수 있다. 그 매커니즘은 위와 같지만, 서로 분리되어 있던 mRNA이기 때문에 고리모양(ρ[8]) 대신 Y자 모양의 인트론이 형성된다.
각주
- ↑ 아예 없진 않다.
- ↑ 이 이름은 나중에 Walter Gilbert에 의해 붙여진 것으로, 처음에는 split gene이라고 불렸다.
- ↑ Gilbert, Walter (1978). "Why genes in pieces". Nature 271 (5645): 501–501.
- ↑ "Physiology or Medicine 1993 - Press Release". Nobelprize.org. Nobel Media AB 2014. Web. 27 Nov 2015. [1]
- ↑ Padgett RA, Grabowski PJ, Konarska MM, Seiler S, Sharp PA (1986). "Splicing of messenger RNA precursors". Annu. Rev. Biochem. 55: 1119–50
- ↑ Breathnach, R. and Chambon, P. (1981) Organization and expression of eucaryotic split genes coding for proteins. Annu. Rev. Biochem. 50, 349–383
- ↑ A는 이미 branch site에서 두개의 염기와 결합한 상태라는 것을 상기해보자.
- ↑ 단순히 모양을 표현한 것일 뿐, 실제 저 기호의 의미와는 관련 없음